R4CR
Day2 - tableone | 2023-09-01
Jinseob Kim
시작하기 전에
본 자료는 데이터셋의 변수를 하나의 테이블로 요약하는 방법에 대해 알아볼 것이다. tableone 패키지를 이용하면 효율적으로 논문에 들어갈 table1을 만들 수 있다.
What is tableone?
tableone은 의학 연구 논문에서 볼 수 있는 table1을 구성하는 데 사용되는 패키지로, 한 테이블 내에 혼합된 범주형 변수와 연속형 변수를 모두 요약할 수 있고 사용법이 매우 간단하다는 장점이 있다.
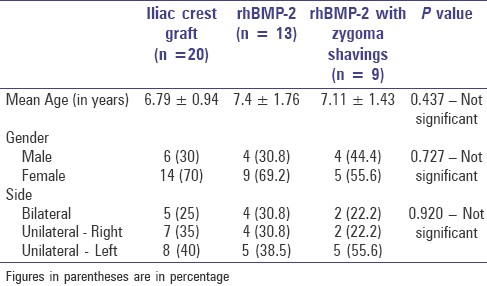
Creating a tableone
CreateTableOne() 함수를 사용하여 테이블을 만들 수 있다. fread를 통해 데이터를 불러와 간단한 테이블을 만들어보자. 데이터는 09-15년 공단 건강검진 데이터에서 실습용으로 32명을 뽑은 자료이며, 자세한 내용은 용어-건강보험데이터를 참고하자.
tableone 2
tableone 3 +
CreateTableOne() 함수를 사용해 간단한 테이블을 만들어 보자.
- 범주형 변수는 개수(백분율)로 요약된다.
- 연속형 변수는 normal인 경우 mean(sd)로 나타나고, nonnormal인 경우 median(IQR)로 요약된다.
| Overall | |
|---|---|
| n | 1644 |
| EXMD_BZ_YYYY (mean (SD)) | 2012.08 (1.99) |
| RN_INDI (mean (SD)) | 490782.25 (290056.68) |
| HME_YYYYMM (mean (SD)) | 201216.18 (199.11) |
| Q_PHX_DX_STK (mean (SD)) | 0.01 (0.11) |
| Q_PHX_DX_HTDZ (mean (SD)) | 0.02 (0.15) |
Argument customize
CreateTableOne() 함수에는 다양한 옵션이 존재한다. 세부적인 옵션 설정을 통해 원하는 table1을 만들 수 있다. 주요 옵션은 다음과 같다.
CreateTableOne- vars : 테이블에 들어갈 변수들
- factorVars : 범주형 변수들
- data : 데이터
- strata : 그룹 변수 지정
- includeNA : 범주형 변수에서
NA를 하나의 범주로 포함할지 여부
옵션에 대한 자세한 설명은 예시를 통해 다루도록 하겠다.
Categorical variable conversion +
factorVars 인자를 사용하여 범주형 변수를 지정할 수 있다. 이때 vars 인자를 통해 전체 데이터 셋 중 테이블에 들어갈 변수를 설정할 수 있고, 지정하지 않을 시 데이터 셋의 모든 변수가 포함된다.
| Overall | |
|---|---|
| n | 1644 |
| HGHT (mean (SD)) | 164.55 (9.19) |
| WGHT (mean (SD)) | 65.10 (12.53) |
| BMI (mean (SD)) | 23.97 (3.39) |
| Q_PHX_DX_STK = 1 (%) | 12 (1.1) |
| Q_PHX_DX_HTDZ = 1 (%) | 26 (2.4) |
Categorical variable conversion +
범주형 변수로 설정한 컬럼의 요약값이 mean(sd)에서 n(percentage)로 바뀐 것을 볼 수 있다.
두 개의 범주가 있는 범주형 변수의 경우, 두 번째 범주의 요약값만 출력된다. 예를 들어 0과 1의 범주가 있을 때, 범주1의 개수와 백분율이 출력된다. 이는 옵션 설정을 통해 전체 범주의 요약값을 출력하도록 변경할 수 있다.
3개 이상의 범주가 있을 때에는 모든 범주의 값이 요약되며, 백분율은 누락된 값을 제외한 후 계산된다.
Multiple group summary +
strata 인자를 설정하여 그룹별 연산을 할 수 있다. strata는 dplyr 패키지의 group_by() 함수와 유사하며, 그룹 연산을 할 변수를 지정하여 사용할 수 있다.
| 1 | 2 | 3 | p | test | |
|---|---|---|---|---|---|
| n | 995 | 256 | 391 | ||
| HGHT (mean (SD)) | 160.67 (8.34) | 168.83 (6.45) | 171.61 (7.09) | <0.001 | |
| WGHT (mean (SD)) | 61.17 (11.08) | 70.09 (10.72) | 71.76 (13.07) | <0.001 | |
| BMI (mean (SD)) | 23.68 (3.41) | 24.57 (2.95) | 24.31 (3.52) | <0.001 | |
| Q_PHX_DX_STK = 1 (%) | 11 (1.8) | 1 (0.5) | 0 (0.0) | 0.051 | |
| Q_PHX_DX_HTDZ = 1 (%) | 18 (2.9) | 5 (2.6) | 3 (1.1) | 0.287 |
Multiple group summary +
연속형 변수의 경우, 기본적으로 one-way ANOVA test가 적용되며 nonnormal일 경우 옵션 설정을 통해 Kruskal–Wallis one-way ANOVA test를 적용할 수 있다.
범주형 변수의 경우, 기본적으로 chisq-test가 적용되며 print 함수의 exact 옵션 설정을 통해 fisher-test를 적용할 수 있다.
Print tableone
CreateTableOne() 함수를 사용하여 테이블을 만든 후, print 명령어로 세부 옵션을 지정할 수 있다. 주요 옵션은 다음과 같다.
print- showAllLevels, cramVars : 2범주인 변수에서 2범주를 다 보여줄 변수
- nonnormal : 비모수통계를 쓸 연속 변수
- exact : fisher-test를 쓸 범주형 변수
- smd : standardized mean difference
Showing all levels
범주형 변수에서 모든 범주의 요약값을 확인하려면 ShowAllLevels 또는 cramVars 옵션을 사용한다. ShowAllLevels = T 를 설정하거나 cramVars 옵션에 원하는 변수명을 지정하여 사용할 수 있다.
showAllLevels +
| level | Overall | |
|---|---|---|
| n | 1644 | |
| HGHT (mean (SD)) | 164.55 (9.19) | |
| WGHT (mean (SD)) | 65.10 (12.53) | |
| BMI (mean (SD)) | 23.97 (3.39) | |
| Q_PHX_DX_STK (%) | 0 | 1059 (98.9) |
| 1 | 12 ( 1.1) | |
| Q_PHX_DX_HTDZ (%) | 0 | 1052 (97.6) |
| 1 | 26 ( 2.4) |
cramVars +
| Overall | |
|---|---|
| n | 1644 |
| HGHT (mean (SD)) | 164.55 (9.19) |
| WGHT (mean (SD)) | 65.10 (12.53) |
| BMI (mean (SD)) | 23.97 (3.39) |
| Q_PHX_DX_STK = 0/1 (%) | 1059/12 (98.9/1.1) |
| Q_PHX_DX_HTDZ = 1 (%) | 26 (2.4) |
nonnormal variables +
비모수통계를 사용하는 연속형 변수에는 nonnormal 옵션을 설정한다. nonnormal 설정 시 mean(sd)에서 median(IQR)로 요약값이 변경된다.
| Overall | |
|---|---|
| n | 1644 |
| HGHT (mean (SD)) | 164.55 (9.19) |
| WGHT (mean (SD)) | 65.10 (12.53) |
| BMI (mean (SD)) | 23.97 (3.39) |
| Q_PHX_DX_STK = 1 (%) | 12 (1.1) |
| Q_PHX_DX_HTDZ = 1 (%) | 26 (2.4) |
exact +
exact 옵션을 통해 fisher-test를 진행할 범주형 변수를 설정할 수 있다. 범주형 변수는 기본적으로 chisq-test가 적용되며, exact 옵션에 fisher-test를 적용할 변수를 지정하여 사용할 수 있다.
| 1 | 2 | 3 | p | test | |
|---|---|---|---|---|---|
| n | 995 | 256 | 391 | ||
| HGHT (mean (SD)) | 160.67 (8.34) | 168.83 (6.45) | 171.61 (7.09) | <0.001 | |
| WGHT (mean (SD)) | 61.17 (11.08) | 70.09 (10.72) | 71.76 (13.07) | <0.001 | |
| BMI (mean (SD)) | 23.68 (3.41) | 24.57 (2.95) | 24.31 (3.52) | <0.001 | |
| Q_PHX_DX_STK = 1 (%) | 11 (1.8) | 1 (0.5) | 0 (0.0) | 0.045 | exact |
| Q_PHX_DX_HTDZ = 1 (%) | 18 (2.9) | 5 (2.6) | 3 (1.1) | 0.281 | exact |
smd +
smd 옵션을 통해 smd(standardized mean difference)를 table1에 포함할 수 있다. default는 FALSE이고, smd=TRUE 설정 시 각 변수의 smd 값이 출력된다.
| 1 | 2 | 3 | p | test | SMD | |
|---|---|---|---|---|---|---|
| n | 995 | 256 | 391 | |||
| HGHT (mean (SD)) | 160.67 (8.34) | 168.83 (6.45) | 171.61 (7.09) | <0.001 | 0.972 | |
| WGHT (mean (SD)) | 61.17 (11.08) | 70.09 (10.72) | 71.76 (13.07) | <0.001 | 0.611 | |
| BMI (mean (SD)) | 23.68 (3.41) | 24.57 (2.95) | 24.31 (3.52) | <0.001 | 0.180 | |
| Q_PHX_DX_STK = 1 (%) | 11 (1.8) | 1 (0.5) | 0 (0.0) | 0.051 | 0.137 | |
| Q_PHX_DX_HTDZ = 1 (%) | 18 (2.9) | 5 (2.6) | 3 (1.1) | 0.287 | 0.084 |
Detailed information : summary()
summary 함수를 쓰면 누락값을 포함한 table1의 자세한 정보를 알 수 있다. 연속형 변수의 값들이 먼저 출력되며 그 다음으로 범주형 변수의 요약값이 출력된다.
### Summary of continuous variables ###
strata: Overall
n miss p.miss mean sd median p25 p75 min max skew kurt
HGHT 1644 0 0 165 9 165 158 171 134 188 -0.2 -0.3
WGHT 1644 0 0 65 13 64 56 73 31 118 0.7 1.0
BMI 1644 0 0 24 3 24 22 26 12 37 0.4 0.4
=======================================================================================
### Summary of categorical variables ###
strata: Overall
var n miss p.miss level freq percent cum.percent
Q_PHX_DX_STK 1644 573 34.9 0 1059 98.9 98.9
1 12 1.1 100.0
Q_PHX_DX_HTDZ 1644 566 34.4 0 1052 97.6 97.6
1 26 2.4 100.0
Show categorical or continuous variable only +
앞에서 만든 table1에서 연속형 변수와 범주형 변수를 구분하여 출력할 수 있다. “$”를 사용하여 연속형 변수는 ConTable, 범주형 변수는 CaTable을 설정하면 해당 변수를 따로 출력할 수 있다.
| 1 | 2 | 3 | p | test | |
|---|---|---|---|---|---|
| n | 995 | 256 | 391 | ||
| Q_PHX_DX_STK = 1 (%) | 11 (1.8) | 1 (0.5) | 0 (0.0) | 0.051 | |
| Q_PHX_DX_HTDZ = 1 (%) | 18 (2.9) | 5 (2.6) | 3 (1.1) | 0.287 |
Categorical Only +
| 1 | 2 | 3 | p | test | |
|---|---|---|---|---|---|
| n | 995 | 256 | 391 | ||
| HGHT (mean (SD)) | 160.67 (8.34) | 168.83 (6.45) | 171.61 (7.09) | <0.001 | |
| WGHT (mean (SD)) | 61.17 (11.08) | 70.09 (10.72) | 71.76 (13.07) | <0.001 | |
| BMI (mean (SD)) | 23.68 (3.41) | 24.57 (2.95) | 24.31 (3.52) | <0.001 |
Export tableone
write.csv 함수를 사용하여 table1을 csv파일로 저장할 수 있다. write.csv(x, file=“파일명”) 형식을 사용하여 csv파일로 저장한다.
마치며
이번 강의를 정리하자.
tableone 패키지의
CreateTableone함수를 사용하여 요약 통계량 테이블을 작성할 수 있다.CreateTableone함수에는 다양한 옵션이 존재하며 strata 옵션을 통해 그룹별 통계량을 계산할 수 있다.CreateTableOne함수를 사용하여 테이블을 만든 후, print 명령어로 세부 옵션을 지정할 수 있다.summary함수를 쓰면 누락값을 포함한 table1의 자세한 정보를 알 수 있다.write.csv함수를 사용하여 tableone 패키지로 만든 table을 csv파일로 저장할 수 있다.
