R4CR
Day1 - tidyverse2 | 2023-06-19
Jinhwan Kim
Overview
Data management with Tidyverse
데이터를 읽고 readr
데이터를 정리하고 dplyr
통계 분석을 실행하고 purrr (워크샵에선 안 다룸)
결과를 만들고 ggplot2
외부에 공유 rmarkdown, quarto, shiny
몰라도 baseR로 할 수 있지만, 알면 편해진다.
readr
- (로컬의) 파일을 (R로) 읽어오는 용도
- 주요 파라미터
- file, col_names
- col_types.
character,integer,number,double,logical,factor,Date,time,_,-: Skip - na: NA로 처리할 글자.
,na,999,-… - col_select : 읽을 column (
select) - skip
- n_max

readr & 3 Core function
read_csv(): Comma(,)로 구분된 데이터
read_tsv(): Tab(\t) 으로 구분된 데이터
read_table(): 공백( ) 으로 구분된 데이터
대신 쓸 수 있는 방법
- base R: read.csv(), read.delim(), read.table()
- data.table : fread()
base R vs readr
- 보통 10~100배 빠른 속도
- col_names, col_types vs header, colClasses
- date / time을 인식
- Progress bar (진행 상황 ::…)
- read_csv() vs fread()
- 데이터가 클 수록 fread()가 빠름
- delimiter, skipped row, header를 설정하지 않아도 됨
- tidyverse (tibble) 과의 연계
dplyr
- d(ata)-plyr
- 기록 시점의 데이터와 분석에 필요한 데이터는 다름
- 필요하지 않은 데이터
- (지표등을) 계산, 추가해야 하는 경우
library(dplyr)
starwars %>%
filter(species == "Droid") %>%
mutate(bmi = mass / ((height / 100) ^ 2)) %>%
select(name:mass, bmi) %>% # name 에서 mass 까지 + bmi
arrange(desc(mass)) # Decrease. desc는 %>%로 안쓰는 것이 좋음
starwars %>%
group_by(species) %>%
summarise(
n = n(), # count
mass = mean(mass, na.rm = TRUE)
) %>%
filter(
n > 1,
mass > 50
)
dplyr & 5 Core function
mutate(): 새로운 Column 추가
select(): 조건에 따라 Column 선택
filter(): 조건에 따라 Row 선택 (base R의 subset과 비슷)
group_by() & summarise(): 요약 (통계치) 계산
arrange(): Row 순서 변경 (정렬)
dplyr - filter
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
1 5.1 3.5 1.4 0.2 setosa
2 5.0 3.6 1.4 0.2 setosa
3 5.4 3.9 1.7 0.4 setosa
4 5.4 3.7 1.5 0.2 setosa
5 5.8 4.0 1.2 0.2 setosa
6 5.7 4.4 1.5 0.4 setosa Sepal.Length Sepal.Width Petal.Length Petal.Width Species
1 5.1 3.5 1.4 0.2 setosa
2 5.0 3.6 1.4 0.2 setosa
3 5.4 3.9 1.7 0.4 setosa와는 결과가 다름
dplyr - multiple filter
dplyr - arrange (sort)
dplyr - arrange 2
dplyr - select
dplyr - select 2
- 제외
- 범위 선택
dplyr - mutate
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
1 5 3.5 1.4 0.2 setosa
2 5 3.0 1.4 0.2 setosa
3 5 3.2 1.3 0.2 setosa
4 5 3.1 1.5 0.2 setosa
5 5 3.6 1.4 0.2 setosa
6 5 3.9 1.7 0.4 setosadplyr - group by & summarise
보통 둘이 같이 씀
baseR의 aggregation과 유사한 목적
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
1 5.1 3.5 1.4 0.2 setosa
2 4.9 3.0 1.4 0.2 setosa
3 4.7 3.2 1.3 0.2 setosa
4 4.6 3.1 1.5 0.2 setosa
5 5.0 3.6 1.4 0.2 setosa
6 5.4 3.9 1.7 0.4 setosa[1] 5.006dplyr - inner_join
baseR의 merge와 유사한 목적
by로 공통 column 설정 merge에서 all.X & all.Y로 남길 값 선택했는데 여기서는 left_join, right_join으로 사용
추가 자료: across
column들을 프로그래밍적으로 선택

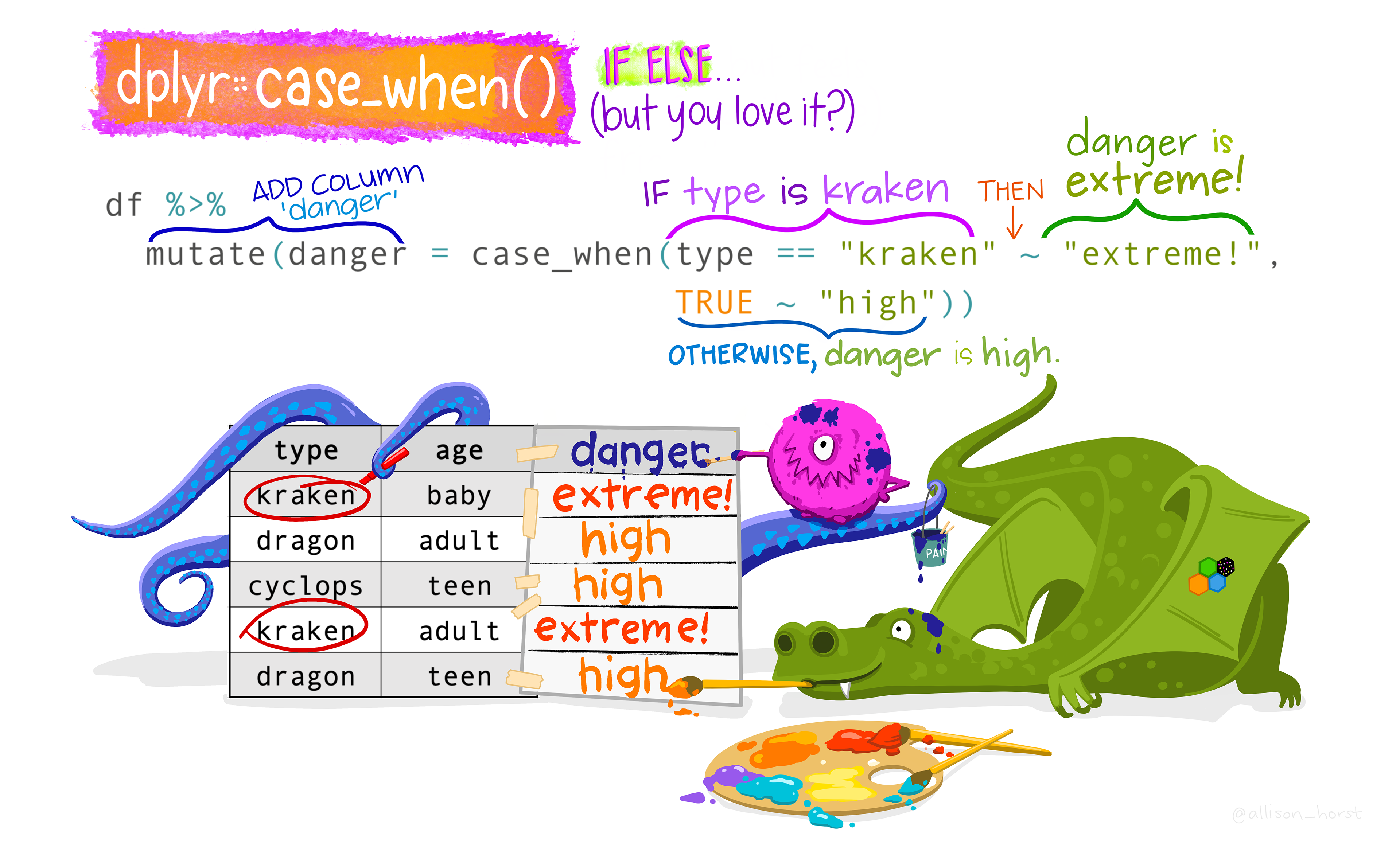
추가 자료: case_when
ifelse와 유사
추가 자료: mutate

추가 자료: relocate
column의 순서 변경

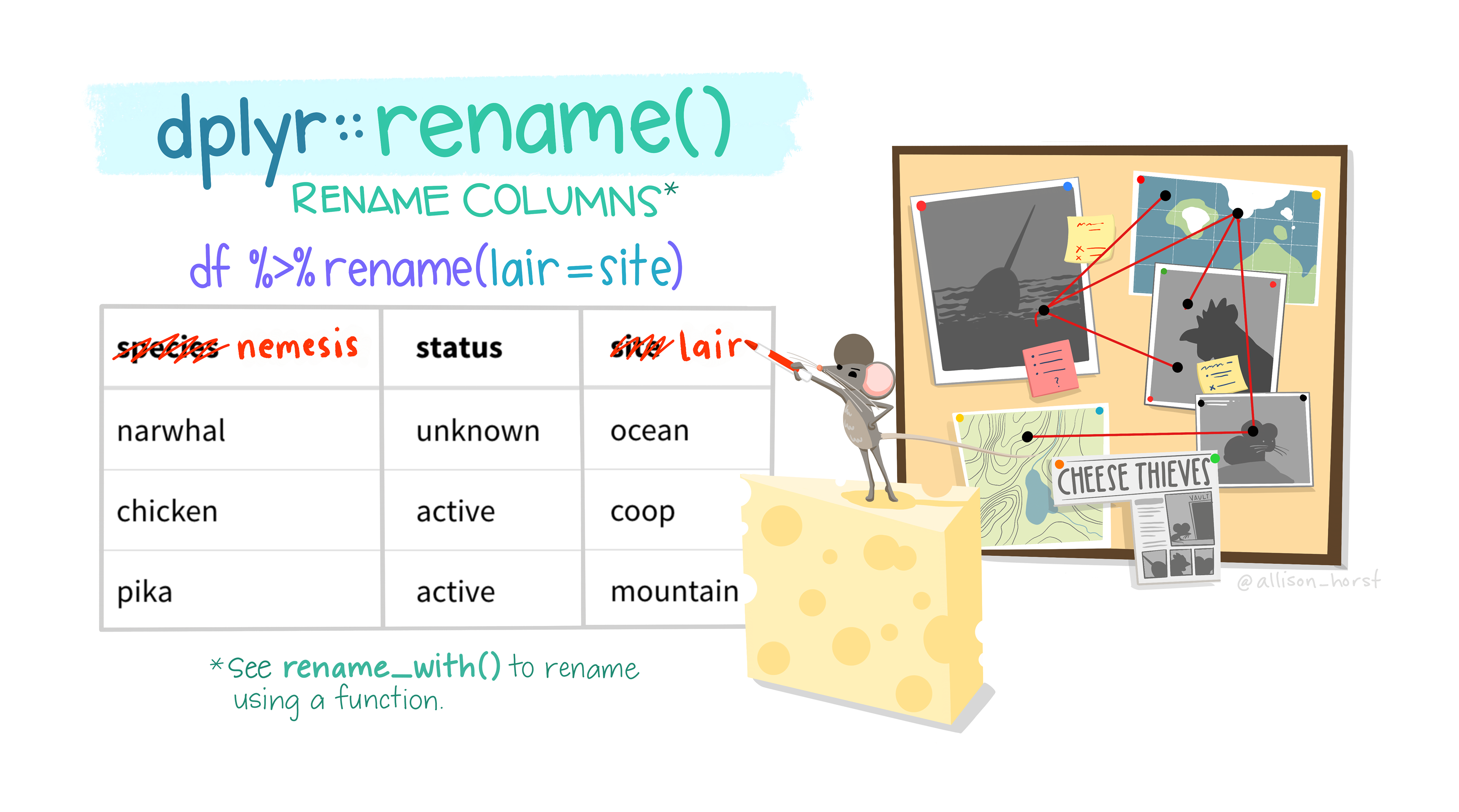
추가 자료: rename
column의 이름 변경. 자주 씀
정리
dplyr는 %>%와 함께 데이터를 변환하는 패키지
5개의 주요 함수는 익숙해지면 좋음.
- mutate
- select
- filter
- arrange
- group_by & summarise
더 많은 정보: Introduction to dplyr 아티클
추가자료의 일러스트는 Allison Horst의 작품
- dplyr외에도 다른 좋은 일러스트도 많음 !


